El descubrimiento y la comercialización de un nuevo fármaco sigue siendo un proceso largo y arriesgado: de 10 a 15 años de media, con una elevada tasa de fracaso y un coste medio de 2.000 millones de euros. “Durante la fase de descubrimiento, hay etapas de exploración para comprender la patología que buscamos tratar e identificar una diana terapéutica, luego una fase de cribado de diferentes moléculas con vistas a modular la actividad de la diana. Sólo entonces entramos en la fase de pruebas preclínicas y luego clínicas”, explica Jeremy Grignard, investigador y científico de datos del Instituto de Investigación Servier. Por lo tanto, uno de los desafíos para los laboratorios farmacéuticos es aumentar la tasa de éxito de los candidatos a fármacos que progresarán a través de todas las fases clínicas en humanos.
Precisamente sobre este tema, el laboratorio Servier, especializado en enfermedades cardiovasculares, cánceres, diabetes, enfermedades inmunoinflamatorias y neuropsiquiátricas, acogió a Jeremy Grignard para una tesis de Cifre en 2017, en colaboración con Inria-Saclay. Continuando con su trabajo, el Instituto de Investigación del grupo creó un departamento de ciencia de datos en 2022, en particular para explorar el potencial de las tecnologías de aprendizaje automático y profundo.
Integrar conceptos muy heterogéneos en un mismo modelo
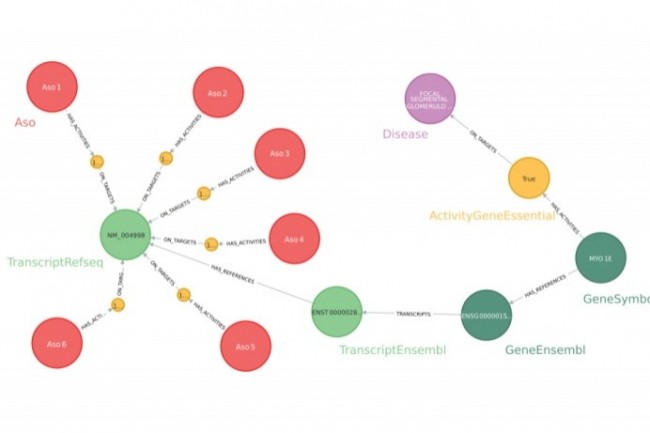
En total, los investigadores farmacéuticos trabajan con casi cincuenta fuentes de datos diferentes durante las fases de descubrimiento. “Aparte de este volumen tan importante, los conceptos farmacobiológicos son muy heterogéneos. El proyecto Pegasus tenía como objetivo integrar y federar todos estos datos en un gráfico de conocimiento. Incluye algo más de 46 millones de entidades con 66 etiquetas diferentes y más de 330 millones de relaciones, de 14 tipos diferentes”, testifica el científico de datos. Para construir gráficos, eligió trabajar con Neo4J, tanto por la flexibilidad del modelo de datos, que cumplió con los desafíos de escalabilidad en el tiempo, como por sus eficientes capacidades de almacenamiento y consulta, así como por su fácil preparación de datos. "Pegasus fue diseñado para proporcionar respuestas muy rápidamente, con el fin de ayudar a los investigadores a probar y comparar diferentes hipótesis", explica Jeremy Grignard. “Responder a una pregunta científica determinada significa recorrer una serie de caminos en el gráfico del conocimiento. »
Pegasus funciona con datos sin procesar, que provienen de bases de datos científicas o de modelos de inteligencia artificial. Luego, los usuarios acceden a él a través de aplicaciones e informes analíticos automatizados. Entre los primeros casos de uso explorados se encuentra la búsqueda de oligonucleótidos antisentido (ASO) que permitan modular una proteína diana. “De 100.000 ASO diseñados para un objetivo terapéutico, priorizamos 784 que validamos experimentalmente, comprobando, por ejemplo, aquellos que corrían el riesgo de tener actividades sobre objetivos distintos a los definidos, para ser excluidos debido al riesgo de "efectos secundarios", ilustra Jeremy Grignard.
Una tasa de acierto del 15%
Históricamente, la selección de moléculas químicas se realizaba de forma aleatoria entre millones de candidatos. En algunos experimentos, se comparó la solución basada en gráficos de conocimiento con los resultados obtenidos mediante el proceso clásico, para observar si permitía enriquecer el número de aciertos. "Identificamos 984 moléculas con una tasa de acierto del 15%, mientras que por defecto ronda el 1%", indica Jeremy Grignard. La solución se utiliza ahora en varios proyectos terapéuticos, con el fin de aumentar la tasa de éxito y la calidad de las moléculas seleccionadas.

Otras noticias que te pueden interesar